YaDiV
 YaDiV ("Yet Another DIcom Viewer") ist ein Programm zur interaktiven Visualisierung von 3D Volumendaten aus dem Bereich der Medizin. Das Programm kann Daten im DICOM Format (genaugenommen DICOM File Sets) einlesen und enthält u.a. Module zur
YaDiV ("Yet Another DIcom Viewer") ist ein Programm zur interaktiven Visualisierung von 3D Volumendaten aus dem Bereich der Medizin. Das Programm kann Daten im DICOM Format (genaugenommen DICOM File Sets) einlesen und enthält u.a. Module zur
- 2D Visualisierung,
- 3D Volumen-Visualisierung (2- und 3D-texturbasiert),
- 3D Segmentierung,
- 3D Segment-Visualisierung,
- Unterst√ľtzung f√ľr stereographische Visualisierung und haptische Eingabeger√§te.
Das Programm wird seit 2005 von Dr. Karl-Ingo Friese zusammen mit Studenten des Welfenlab entwickelt und dient derzeit als Plattform f√ľr unsere eigene Forschung im Bereich der medizinischen 3D-Datenverarbeitung. Eine Open Source Ver√∂ffentlichung ist in K√ľrze geplant!
Download
Die f√ľnfte Beta-Version von YaDiV ist inzwischen als Closed Source erh√§ltlich! YaDiV 1.0 beta 5 ist nahezu identisch mit der aktuellen Entwickler Version, lediglich die haptische Schnittstelle wurde entfernt.
Download YaDiV 1.0 beta5 (ca. 6MB)
Neu in der 5. Beta Version sind u.a.: Untest√ľtzung weiterer Volumenformate, Deformable Model Segmentierung, Identifikation + Analyse von Zusammenhangskomponenten und vieles mehr.
Diese Beta-Version darf nur zum Testen des Programmes heruntergeladen und nicht weitergegeben werden. Eine nicht-kommerzielle Nutzung im Rahmen von Forschung und Lehre ist erlaubt, n√§here Details stehen in der mitgelieferten Programmhilfe. Das Programm muss nach dem Herunterladen nicht installiert werden. Einfach die zip Datei auspacken und LaunchYaDiV.jar ausf√ľhren. Eine aktuelle Java Version (6.x oder neuer) sowie Java3D (1.5.2) sollten auf dem System bereits installiert sein. Frei erh√§ltliche DIOCM Datens√§tze k√∂nnen bspw. hier heruntergeladen werden. Je nach Aufl√∂sung des Datensatzes werden 2-4 GB Hauptspeicher ben√∂tigt.
Das Programm ist noch nicht fertig, daher kann es vorkommen das DICOM Datens√§tze nicht richtig angezeigt werden oder dass das Programm abst√ľrzt. Benutzer-R√ľckmeldungen (Fehlerberichte, Feature-W√ľnsche) sind daher jederzeit willkommen.
Technisches
YaDiV wird in der Programmiersprache Java entwickelt und l√§uft problemlos unter Windows, Linux (fedora 8) und MacOS X. Das Programm wurde unter 32 und 64 bit getestet, letzteres ist f√ľr sehr gro√üe Volumendaten absolut erforderlich. Da YaDiV speziell f√ľr schnelle, interaktive Visualisierung und Segmentierung angelegt wurde, ist es stark thread-basiert und profitiert direkt von Mehrkernprozessoren bzw. Multiprozessorumgebungen. Aber auch auf einem normalen Laptop lassen sich mittlere Voxeldatendatens√§tze (~ 512¬≥) schnell ansehen und bearbeiten.
F√ľr die 3D-Visualisierung wird Java3D (Version 1.5.2) eingesetzt. Alle zeitkritischen Verfahren wie eine Level-Set Segmentierung oder das Einlesen gro√üer Daten wurden als eigener Thread implementiert, so dass die Programmoberfl√§che niemals blockiert wird. So wird z.B. nach einer √Ąnderung eines Segmentes im Hintergrund ein automatisch verfeinernder Algorithmus f√ľr die 3D Darstellung gestartet, damit √Ąnderungen sofort sichtbar werden. Wenn sich das Segment erneut √§ndert bevor die h√∂chste Verfeinerungsstufe erreicht wurde, startet der Verfeinerungsalgorithmus automatisch neu.
Im Zuge der Entwicklung wurden mehrere vom Hauptpaket unabh√§ngige APIs entworfen, z.B. eine Settings-API, die es erlaubt zur Laufzeit spezielle Klassenattribute zu definieren die automatisch mit einem GUI Element verkn√ľpft werden. F√ľr das Layout wurde ebenfalls eine spezielle API ("GridMaker") entworfen, die die Layout Beschreibung der SWING Oberfl√§che in einer HTML-Tabellen √§hnlichen Syntax erlaubt. Beide APIs, GridMaker und die Settings-API werden in naher Zukunft als eigenst√§ndige Open Source Pakete ver√∂ffentlicht.
Screenshots
 |
 |
 |
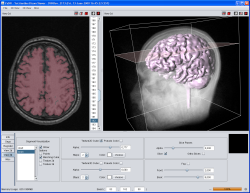
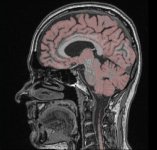
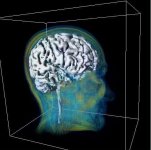
| YaDiV Hauptfenster | Region of Interest | Segmentierung |
 |
 |
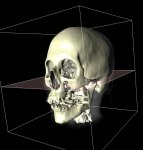 |
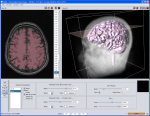
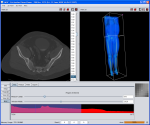
| 2D Segmentvisualierung | 3D Segmentvisualierung | Marching Cube |
 |
 |
 |
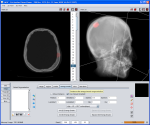
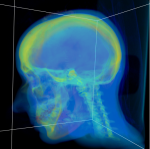
| Pseudo-Color Darstellung | Settings Browser | DICOM File Info Window |
Arbeiten
- Bachelorarbeit Automatische Erkennung des Acetabulum in 3D-Pelvis Modellen. Jurek Leonhardt - in Bearbeitung
- Masterarbeit Entwicklung einer halbautomatischen Segmentierungsmethode zur Bestimmung der intrakraniellen Luftmenge nach einer Schädeloperation. Marco Köhnen - 12/2012
- Diplomarbeit Halbautomatische Segmentierung der knöchernen Strukturen des Kniegelenks in MRT Aufnahmen. Benjamin Fleischer - 05/2012
- Studienarbeit Dynamische Erzeungung von Triangle Strips als Erweiterung des Marching Cube Verfahrens. Friedrich Kr√ľger - 12/2011
- Bachelorarbeit Modelling of realistic Blood Vessel Geometry. Sergi Lazaro - 09/2011
- Bachelorarbeit Untersuchung zur Form- und Mustererkennung auf LC Displays bei benachbarten Farbwerten. Mona Lepke - 09/2011
- Masterarbeit Modellbasierte Orbita Segmentierung und die automatisierte Bestimmung anatomisch relevanter Parameter. Matthias Becker - 04/2011
- Studienarbeit Entwicklung eines Moduls zur automatisierten Bestimmung der Kniegelenkskinematik und -biomechanik durch dreidimensionale in vivo-Analyse mittels MRT. Benjamin Fleischer - 02/2011
- Studienarbeit Illustrative Volumendarstellung mit Lit Sphere Mapping unter Ber√ľcksichtiung von vorhandenen Segmentinformationen. Johannes Wahle - 08/2010
- Diplomarbeit Entwurf und Implementierung eines atlasbasierten Segmentierungsverfahrens f√ľr medizinische Volumendaten. Robert Christian Meyer - 12/2009
- Masterarbeit Entwurf und Implementation einer haptischen Schnittstelle f√ľr YaDiV. Marc Christoph Vollmer - 10/2009
- Seminar Style Transfer Functions for Illustrative Volume Rendering. Johannes Wahle - 09/2009
- Studienarbeit Extraktion von Isoflächen mit Hilfe des Marching-Cubes-Algorithmus. Damaris Schumann - 04/2009
- Masterarbeit High-Quality Ray Casting Verfahren zur Visualisierung segmentierter medizinischer Voxeldaten. Maximilian M√ľller - 04/2009
- Diplomarbeit Komplexe regionenbasierte Segmentierungsverfahren mit der Level Set Methode in der medizinischen Bildverarbeitung. Björn Scheuermann - 09/2008
- Studienarbeit Untersuchung zur Grauwertdarstellung auf LC-Displays. Lara Toma - 06/2008
- Seminar Atlas-based Segmentation. Robert Meyer - 06/2008
- Seminar Three-Dimensional Medical Image Segmentation Using a Graph-Theoretic Energy-Minimisation Approach. Michael Mainik - 06/2008
- Diplomarbeit Analyse von Direct Volume Rendering Verfahren zur Visualisierung von Voxeldaten. Dominik Sarnow - 12/2007
- Masterarbeit Boundary Extraction from Voxel Data Using Contour Lines. Richard Guercke - 11/2007
- Studienarbeit Segmentierung medizinischer Bilddaten mit der Level-Set-Methode. Björn Scheuermann - 10/2007
- Diplomarbeit Algorithmen zur Randflächenapproximation von Volumenkörpern in Voxeldarstellung. Yifan Yu - 02/2007
- Seminar Grauwertdarstellung in medizinischen Anwendungen. Hans-Jörg von Mettenheim - 05/2006


